一周前Cancer Res (IF: 8.378) 新发了一篇文章“LDtrait: An Online Tool for Identifying Published Phenotype Associations in Linkage Disequilibrium”
看文献名很好理解,就是通过输入RS号或者基因组坐标(支持输入一系列RS号、基因组坐标)查询目前已发表的GWAS文献是否有报道输入的位点及高连锁位点显著相关的表型。
如下截图所示:
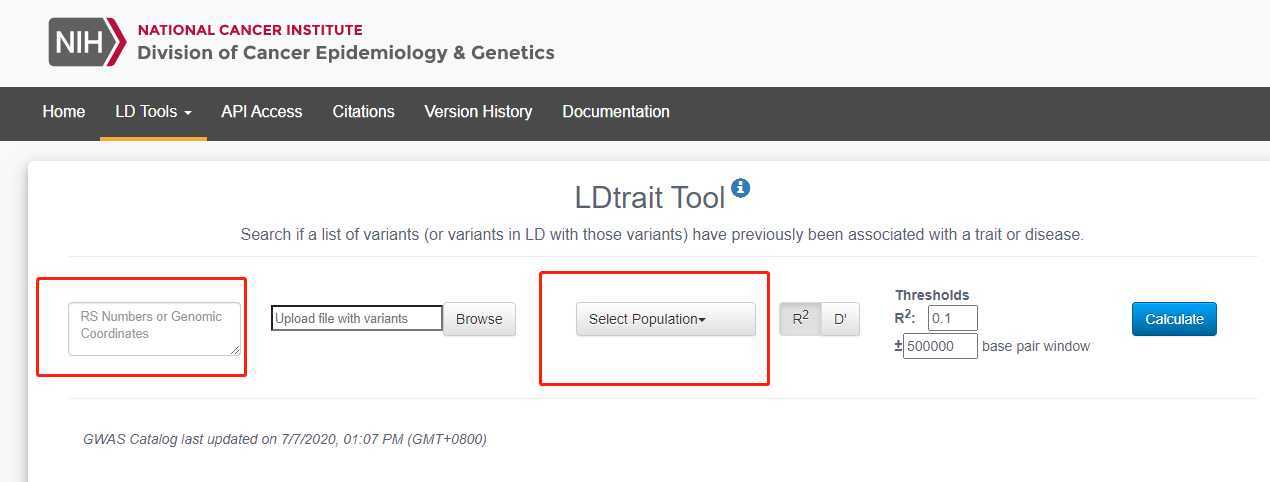
数据调用的是 GWAS Catalog 数据库 (https://www.ebi.ac.uk/gwas/api/search/downloads/alternative),所有GWAS结果的基因组版本均被转化为GRCh37。
新工具有何优点:1)比起之前只能输入单个位点查询数据,该工具支持同时输入多个位点查询数据;2)除了输出查询的位点的相关表型,还会输出查询的位点高连锁的其他的位点的情况。
比如输入位点rs6983267后,就会展示如下的结果:
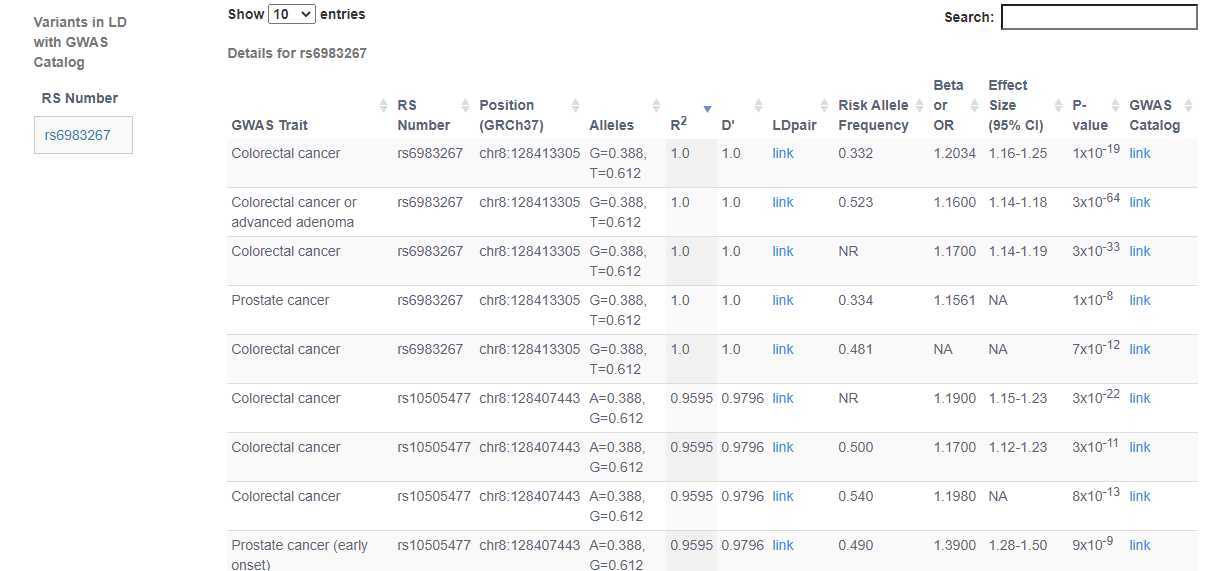
说一下局限吧,该工具调用的是GWAS Catalog,但GWAS Catalog收集的GWAS摘要统计文件不是很齐全。
比如我之前就介绍过数据库“囊括45万人数据,778个表型,3千万个位点的公共数据库:GeneATLAS”,这个数据库就有778个表型,三千万个位点。
LDtrait工具: 查找SNP及其高度连锁位点显著相关的表型
原文:https://www.cnblogs.com/chenwenyan/p/13261537.html