datTraits 是样本特征数据 ,datExpr 是样本表达数据 (这两种数据什么关系) 来自下列数据
femData = read.csv("LiverFemale3600.csv"); datExpr = datExpr0[keepSamples, ] traitData = read.csv("ClinicalTraits.csv"); datTraits = allTraits[traitRows, -1];
先删除后转至改成frame,然后去列名 names () 列:基因 行:样本
WGCNA 关键代码
gsg = goodSamplesGenes(datExpr0, verbose = 3); gsg$allOK #是否全部通过检查 gsg$goodGenes #通过检查的基因 返回数据是 bool 型数组,通过检查的为 true,未通过的为 false gsg$goodSamples #通过检查的样本 返回数据是 bool 型数组,通过检查的为 true,未通过的为 false
sampleTree = hclust(dist(datExpr0), method = "average");
clust = cutreeStatic(sampleTree, cutHeight = 15, minSize = 10)
# Re-cluster samples
sampleTree2 = hclust(dist(datExpr), method = "average")
# 把 Trait 数据转换成颜色,白色表示低,红色表示高,灰表示缺失数据。
traitColors = numbers2colors(datTraits, signed = FALSE);
# Plot the sample dendrogram and the colors underneath.
plotDendroAndColors(sampleTree2, traitColors,
groupLabels = names(datTraits),
main = "Sample dendrogram and trait heatmap")
save(datExpr, datTraits, file = "FemaleLiver-01-dataInput.RData")
lnames = load(file = "FemaleLiver-01-dataInput.RData");#参见:1.2.4
#加载上一节已经预处理的数据。lnames 包含 datExpr 和 datTrait
1.3.3. 选择软阈值
# Choose a set of soft-thresholding powers powers = c(c(1:10), seq(from = 12, to=20, by=2)) #这里的原因有往下的显示 # Call the network topology analysis function sft = pickSoftThreshold(datExpr, powerVector = powers, verbose = 5) # Plot the results: sizeGrWindow(9, 5) par(mfrow = c(1,2)); cex1 = 0.9; # Scale-free topology fit index as a function of the soft-thresholding power plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2], xlab="Soft Threshold (power)",ylab="Scale Free Topology Model Fit,signed R^2",type="n", main = paste("Scale independence")); text(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2], labels=powers,cex=cex1,col="red"); # this line corresponds to using an R^2 cut-off of h abline(h=0.90,col="red") # Mean connectivity as a function of the soft-thresholding power plot(sft$fitIndices[,1], sft$fitIndices[,5], xlab="Soft Threshold (power)",ylab="Mean Connectivity", type="n", main = paste("Mean connectivity"))
左图表示拟合优度 R2 和软阈值之间的关系,红色线表示优度为 0.9,大于 0.9 的说明拟合满意。可选
择 6 作为阈值(无尺度网络参数选择原则 page 错误:引用源未找到)。右图表示平均连接数和软阈值之间
的关系。
blockwiseModules 用于自动网络分析和模块选择。我们这样调用 blockwiseModules。
net = blockwiseModules(datExpr, power = 6, TOMType = "unsigned", minModuleSize = 30, reassignThreshold = 0, mergeCutHeight = 0.25, numericLabels = TRUE, pamRespectsDendro = FALSE, saveTOMs = TRUE, saveTOMFileBase = "femaleMouseTOM", verbose = 3)
datExpr 是数据集。power=6 设置阈值为 6。TOMtype="unsigned" 设置 TOM 矩阵为无符号。
minModuleSize 设置模块选择最小的基因数量为 30。 numericLabels = TRUE 返回的数据包含每个基
因的颜色编号reassignThreshold 设置 p 值的阈值,不满足此阈值时会尝试重新分配基因到其他模块中。
执行完 blockwiseModules 后 net 保存了处理结果。net$colors 保存了 基因根据模块的着色。
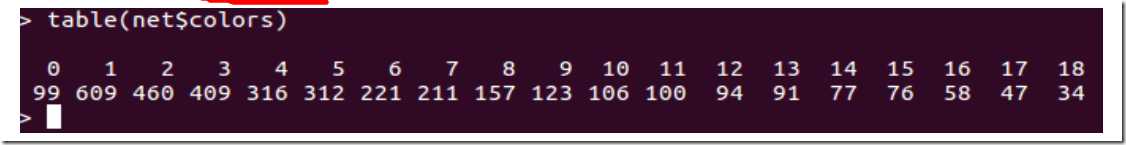
以上命令进行了频数统计。可以发现共有 18 个模块。99 到 34 是每个模块的基因数量。0 代表无法分类
到这 18 个模块当中的基因数量。net$dendrograms[[1]] 保存了用于模块识别的分层聚类图。
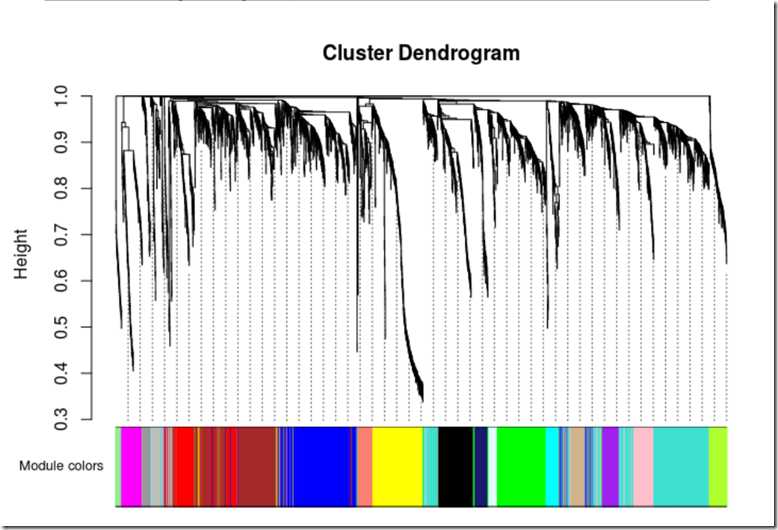
打开图型窗口 sizeGrWindow(12, 9) # 把模块编号转换成颜色值 mergedColors = labels2colors(net$colors) #绘制聚类图和色彩 plotDendroAndColors(net$dendrograms[[1]], mergedColors[net$blockGenes[[1]]], "Module colors", dendroLabels = FALSE, hang = 0.03, addGuide = TRUE, guideHang = 0.05)
原文:https://www.cnblogs.com/mohuishou-love/p/10708955.html