顾名思义,对数据的变量或者观测进行分类。
常见的分类方法有:层次法(凝聚式、分裂式适合,适合观测少的,不需要输入类别数)、划分法(开始阶段直接指定某几个类中心,适合观测多的情形,需要输入类别数)。
检测分类好坏的标准:同一类的相似,不同类的几乎不具备相似性(殊途殊归,同途同归)。
一般情况下使用距离函数来表示两个对象的相似度。好的距离定义要满足三个条件:①D(X,Y)=D(Y,X)②若X!=Y,D(X,Y)>0,否则D(X,Y)=0③D(X,Y)<=D(X,Z)+D(Z,Y)。
PROC DISTANCE过程可以用来计算距离:
PROC DISTANCE DATA=STOCK METHOD=DCORR OUT=DISTDCORR;
VAR INTERVAL(DIV_2010 DIV_2011 DIV_2012 DIV_2013);
RUN;
METHOD=指定距离的计算方法,VAR INTERVAL()表示都是等距变量,具体可查阅帮助文档。
K均值聚类法
步骤:
①选定种子
②读观测,计算于种子的距离
③根据现有的类中的观测中心计算种子(类中心)
④重复②③,直到收敛确定K类种子
⑤重新读观测并分类
PROC FASTCLUS DATA=EX.FOOD_CAL MAXC=5 MAXITER=10 OUT=CLUS; VAR KCAL FAT PROTEIN; RUN;
MAXC=5指定分类数,MAXITER=10指定分类迭代的次数,结果如下:
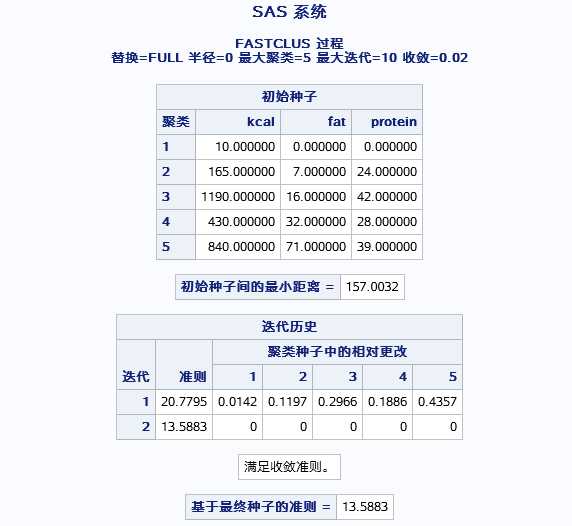
表一为5个中心左表,表二为迭代历史,表三为最终确定的种子为准则为第二次迭代的结果。
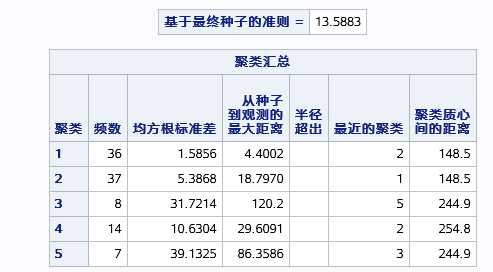
各个类的情况,
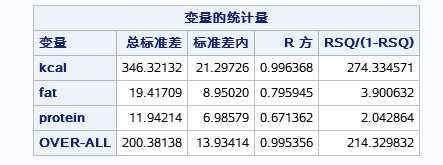
各个变量的统计量,及聚类的标准差等。
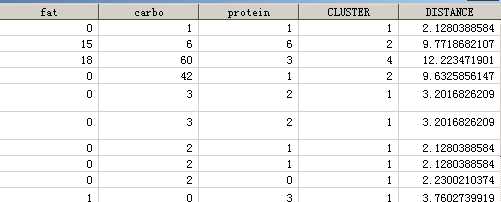
上图是各个观测距离自己类中心的距离值,记录在数据集中。
CLUSTER过程实现层次法分类
代码如下:
PROC CLUSTER DATA=EX.NUTRITION OUTTREE=TREE METHOD=AVE CCC PSEUDO;
VAR MAGNESIUM_MG PERCENT_WATER PROTEIN_G SATURATE_FAT_G;
ID FOOD;
RUN;
METHOD=指定做层次分析的具体方法,自带的方法有11种,上例中用的类平均法。
OUTTREE=指定输出的树数据集,可以用于直接画树形图,
CCC用于输出CCC值,PESUDO用于输出伪F统计量和伪T统计量。
结果包含特征值以及累积解释比例
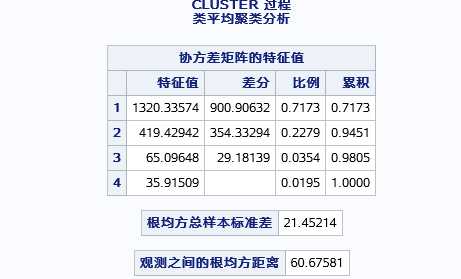
迭代历史仅从倒数几次迭代历史,其中包含几个统计量R方(值越大表示区分的越开,效果越好)、半偏R方(越大,分类效果越不好)、CCC的峰值表示建议聚类数、伪F统计量(越大越好)、伪T统计量(越小分类越合理)。
根据聚类准则,建议分类是3类。
实际上如果cluster过程中包含了outtree选项,系统会自动调用PROC TREE过程来绘画树形图,实际上也可以拿到数据集后自己手动条用proc tree 过程画出树形图。
原文:http://www.cnblogs.com/immaculate/p/6414530.html